Kaum eine Methode hat die Molekularbiologie so sehr verändert wie die Polymerase-Kettenreaktion, kurz PCR. Die Anwendungsbereiche sind schier unüberschaubar geworden und umfassen die Identifikation von Organismen anhand von Gewebeproben (siehe Kurs 1 RFLP), die Identifikation eines Täters / einer Täterin bei einem Verbrechen, einen Vaterschaftsnachweis, die Identifikation von Genen und ihre Sequenzierung, den Nachweis gentechnischer Veränderungen, das Auffinden von Mutationen und die gezielte Veränderung von Gensequenzen, um nur einige Anwendungsbeispiele zu nennen. Die Grundidee ist dabei ebenso einfach wie genial: Mithilfe natürlich vorkommender, hitzestabiler Enzyme, welche DNA vervielfältigen können, kann durch Setzen eines Start- und eines Endpunktes mit Hilfe von speziell designten Oligonucleotiden (sogenannte Primer) sowie durch Zugabe von DNA-Bausteinen (Nukleotiden) bei geeigneten Umgebungsbedingungen eine exponentielle Vervielfältigung der gewünschten DNA-Sequenz in kurzer Zeit erreicht werden.
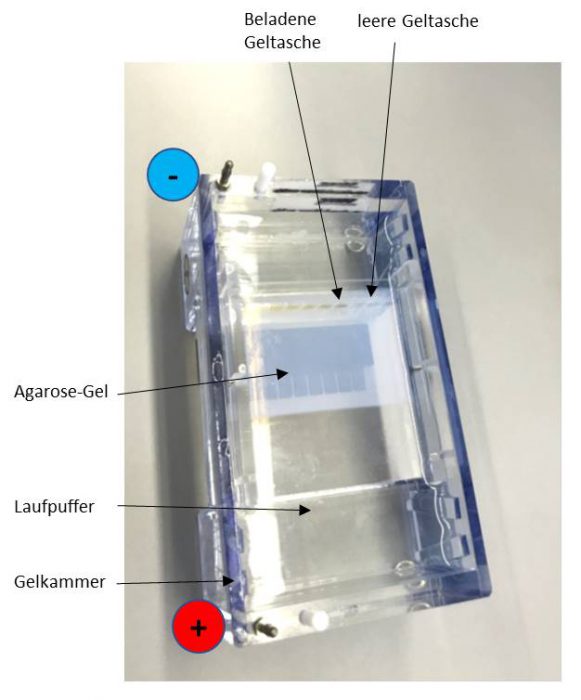
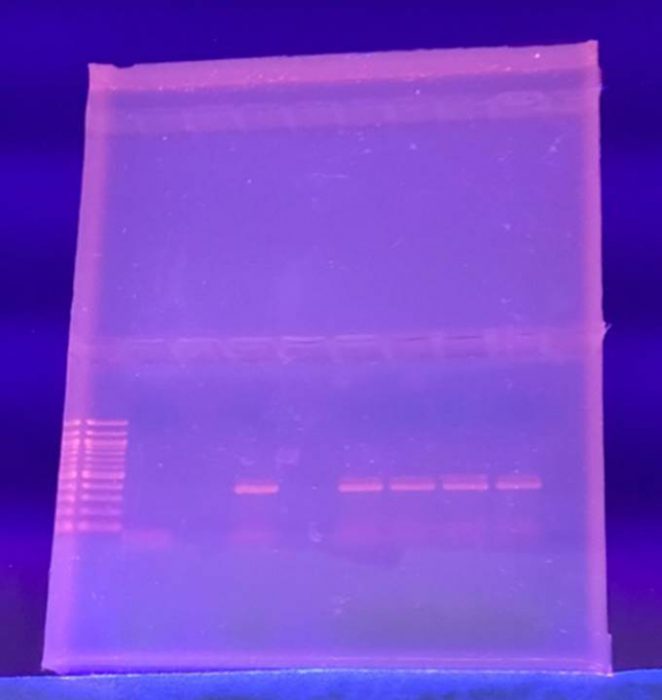
Mit Hilfe der Agarose-Gelelektrophorese können diese PCR-Produkte dann nach Größe aufgetrennt und sichtbar gemacht werden. Hierzu wird üblicherweise ein in die DNA interkalierenden UV-Fluoreszenzmarker wie beispielsweise Ethidiumbromid verwendet. Zur Ermittlung der Größe der Fragmente lässt man gleichzeitig einen sogenannten DNA-Ladder (Marker) mitlaufen, also eine Mischung aus DNA-Fragmenten bekannter Größe. Durch Vergleich dieses MArkers mit der eigenen Probe kann die Größe des PCR-Produktes sowohl grob abgeschätzt als auch näherungsweise genau berechnet werden.
Im Rahmen des Kurses können Schülerinnen und Schüler in Kleingruppen eine PCR durchführen und das Ergebnis auf ein Agarose-Gel übertragen, um den Erfolg der PCR zu überprüfen. Hierbei ergeben sich unterschiedliche Varianten. So kann fertig isolierte DNA verwendet werden, oder es wird selbst DNA isoliert. Die Gelelektrophorese kann selbst durchgeführt werden oder ein hypothetisches Gel ausgewertet werden. Je nach Durchführungsvariante ergeben sich somit unterschiedlich lange Kurszeiten, welche teilweise auch komplett nachmittags außerhalb der regulären Schulzeit stattfinden können. Hier orientieren wir uns soweit möglich an Ihren Wünschen und Möglichkeiten. Material zur Vorbereitung wird in Form von Lernvideos zur Benutzung von Zentrifugen und Kolbenhubpipetten vor dem Kurs zur Verfügung gestellt. Weiterhin stellen wir je nach Kursgestaltung Ablaufpläne vorab zur Verfügung.
Interesse?
Bei Interesse wenden Sie sich bitte an Dr. Sven Dienstbach (dienstbach@biologie.uni-siegen.de, Tel. 02717404575).