Vor einigen Jahren wurde Pferdefleisch in Lasagne gefunden, was sich letztlich zu einem großen Lebensmittelskandal ausweitete. Hier wurde (im Verhältnis zu Rinder- oder Schweinefleisch) billiges Pferdefleisch eingekauft und ohne Deklaration bzw. falsch als Rind bzw. Schwein deklariert, teuer verkauft, um die Gewinnspanne zu erhöhen. Wenn auch aus hygienischer bzw. medizinischer Sicht unproblematisch, so wurde hier doch der Verbraucher arglistig getäuscht, da er in der Regel gerade bei stark verarbeiteten Lebensmitteln kaum eine Möglichkeit hat, zu erkennen, um welches Fleisch es sich ursprünglich gehandelt hat.
Auch außerhalb von Lasagne und stark verarbeiteten Lebensmitteln ist dies jedoch durchaus eine häufige Praxis. Viele Restaurants bieten Seezunge oder Scampi zu vermeintlich günstigen Preisen an. Lebensmitteluntersuchungen zeigen jedoch in erschreckend vielen Fällen, dass es sich bei den Fischen nicht um Seezungen (recht teuer im Einkauf) sondern um andere Plattfische oder gar Pangasiusfilet (Aquakultur, oft ökologisch fragwürdig und teilweise mit Antibiotikabehandlungen) handelt, welche im Einkauf deutlich günstiger sind. Auch bei exotischen Lebensmitteln (Strauß, Känguruh oder Krokodil) kommen entsprechende Täuschungen vor, da Verbrauchern oft die nötige Erfahrung fehlt, wie genau z. B. Straußenfleisch aussieht, welche Textur es hat und welchen Geschmack. Gerade Kängurufleisch ist teilweise im Einkauf sehr günstig zu bekommen und kann in Form von Gulasch auf dem Teller landen, obwohl eigentlich Rindfleisch oder gar Strauß bestellt wurde.
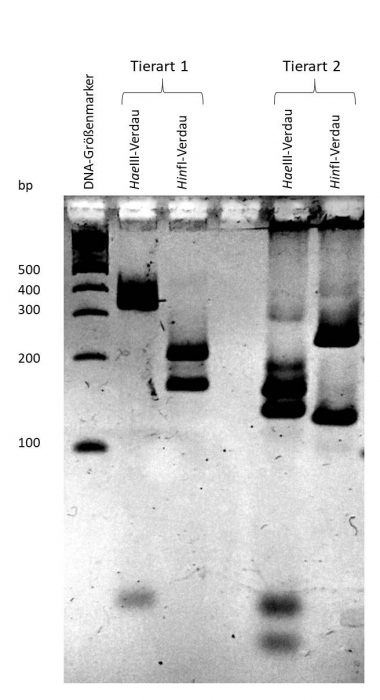
Um solchen Tricksereien auf die Spur zu kommen, kann man eine RFLP-Analyse durchführen. RFLP steht dabei für Restriktions-Fragment-Längen-Polymorphismus. Hierbei wird DNA aus den Proben gewonnen. Danach wird ein bestimmter Abschnitt per PCR (Polymerase-Kettenreaktion) vervielfältigt und anschließend mit ausgewählten Restriktions-Enzymen verdaut. Anhand des Verdaumusters nach Durchführung einer Agarose-Gelelektrophorese kann durch einen Vergleich mit einer entsprechenden Tabelle das in der Probe enthaltene Fleisch eindeutig einer Tierart zugeordnet werden.
Im Rahmen des Kurses erhalten die Schüler in Kleingruppen eine unbekannte Fleischprobe. Sie gewinnen dann aus dieser Probe mittels Säulenpräparation die DNA, führen anschließend eine PCR mit Primern gegen das CytB-Gen durch, prüfen den Erfolg der PCR mittels Agarose-Gelelektrophorese, setzen einen Verdau der PCR-Produkte mit den Enzymen HaeIII und HinfI an, und trennen die Verdaufragmente anschließend über ein Low-melt Agarose-Gel auf. Im Anschluss erfolgt die Identifikation der ausgeteilten Tierart durch Vergleich mit entsprechenden Tabellenwerten. Der Kurs dauert in der Regel von 8 bis 15 Uhr und beinhaltet die Möglichkeit zu einer Mittagspause in der Uni-Mensa. Zur Vorbereitung in der Schule bzw. im Selbststudium werden Lehrvideos zum Umgang mit Zentrifuge und Kolbenhubpipetten zur Verfügung gestellt. Weiterhin stellen wir detaillierte Ablaufpläne vorab zur Verfügung.
Interesse?
Bei Interesse wenden Sie sich bitte an Dr. Sven Dienstbach (dienstbach@biologie.uni-siegen.de, Tel. 02717404575).